
背景介绍
蛋白质互作(Protein Protein Interaction, PPI)是生命活动调控的核心机制。构建蛋白质互作网络能加深对功能基因组学的理解。传统的研究方法很难实现高通量、可靠且成本低廉的蛋白质互作研究,这其中就包括酵母双杂交(yeast two-hybrid, Y2H),荧光共振能量转移(fluorescence resonance energy transfer, FRET)、双分子荧光互补(Bimolecular Fluorescence Complementation, BiFC)等技术。
为研究水稻蛋白质互作组,华中农业大学张建伟团队与中国水稻研究所张健团队联合开发了 BIP-seq(Barcode-indexed PCR Coupled with HiFi Long-read Sequencing)技术,通过整合文库对文库酵母双杂交策略、双条形码索引 PCR 扩增和 HiFi 长读长测序,实现了水稻蛋白互作组的高通量、低成本鉴定。文章以“Global Protein Interactome Mapping in Rice Using Barcode‐Indexed PCR Coupled with HiFi Long‐Read Sequencing”为题于 2025 年发表于《Advanced Science》杂志。
研究方法及结果
研究人员开发的 BIP-seq(Barcode-indexed PCR Coupled with HiFi Long-read Sequencing)技术利用“库对库(library versus library)”的酵母双杂交策略快速获得数以万计的随机的阳性PPI互作组合酵母克隆。接下来通过两轮 Barcode-indexed PCR 将每个酵母克隆中 Bait 和 Prey 载体上的 cDNA 序列直接扩增,并在序列末端添加 2 套条形码用于判断克隆的来源。最后,通过 HiFi long-read 测序方法将数万个扩增子进行混合测序。具有完全相同的 barcode 的 Bait cDNA 和 Prey cDNA 表明它们来自相同的阳性 PPI 互作酵母克隆,即为互作组合(Fig1)。
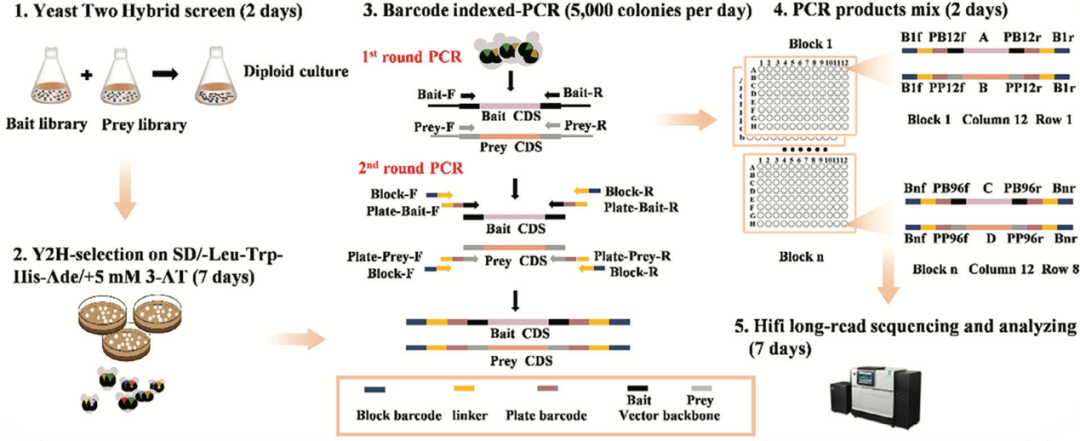
Fig1. The workflow of BIP-seq technology
蛋白互作阳性克隆的挑取,Barcode-indexed PCR 及 HiFi 长读测序
将 Y187(含 bait cDNA 文库)和 Y2HGold(含 prey cDNA 文库)菌株以 0.8mL 等比例混合于 2L 灭菌烧瓶。50mL 2×YPDA 培养基,30℃ 震荡培养 20 小时(50rpm)。杂交后细胞用 15mL 0.9% NaCl 重悬,涂布于四缺培养基(SD/-Leu-Trp-His-Ade,含 5mM 3-AT)。30℃ 培养 7 天后,直径 1-2mm 的菌落判定为潜在蛋白互作阳性。用高通量微生物克隆挑选系统 QPix 420(Fig2)或手动的方式将蛋白互作的酵母菌单克隆挑取到含 10uL 新鲜 KBB 裂解液的 96 孔板中。100℃ 热处理 5min 后即可进行第一轮 Barcode-indexed PCR。
本次实验共设置了 6 个生物学重复,并且每个重复包含 10000 个随机 PPI 克隆。混合 60,000 个 PCR 产物进行 HiFi 长读测序,共获得 7.9 million reads。Barcode-indexed PCR 第一轮和第二轮的效率分别是 87.5%,81.3%。数据过滤后,每重复的有效 bait reads 为 8489.67±595.37,而有效 prey reads 为 8394.5±683.62,匹配的 bait 蛋白数量则为 817.67±105.76,prey 蛋白数量为 2959.83±163.82(Fig3)。

Fig2. QPix420 Colony Picking System
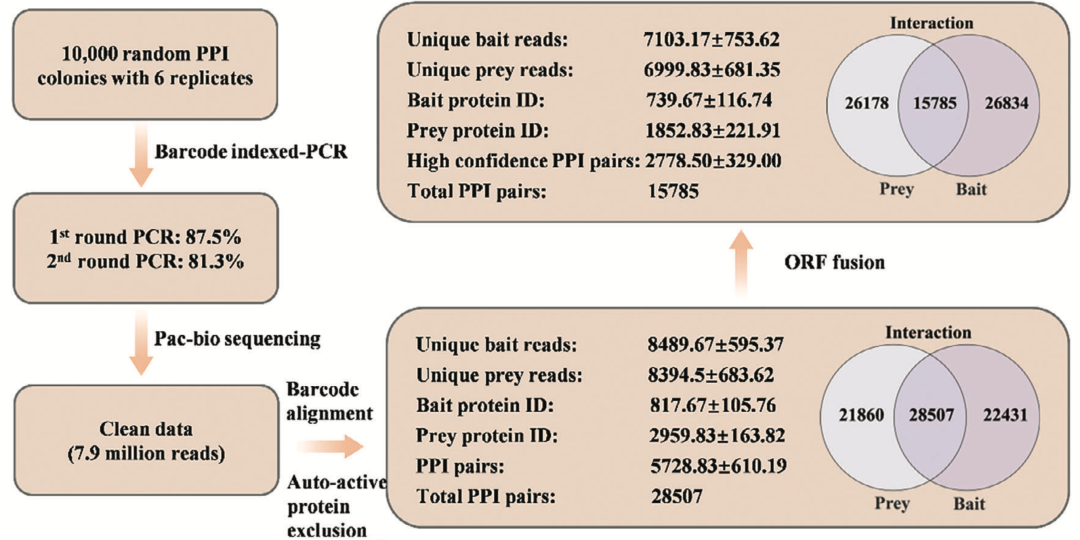
Fig3. The bioinformatic analysis pipeline and output of BIP-seq results on 60000 random PPI colonies
水稻 PPI 数据集
此次水稻 PPI 数据集被命名为 Rice PPI Dataset,即 RiPPID,其中包括 23032 条高置信度互作边(edges)连接 12741 个节点(nodes)(Fig4)。和此前的水稻 PPI 数据集比较,有 22665 条 PPI 是新发现的,极大地扩展了现有的水稻 PPI 数据集。
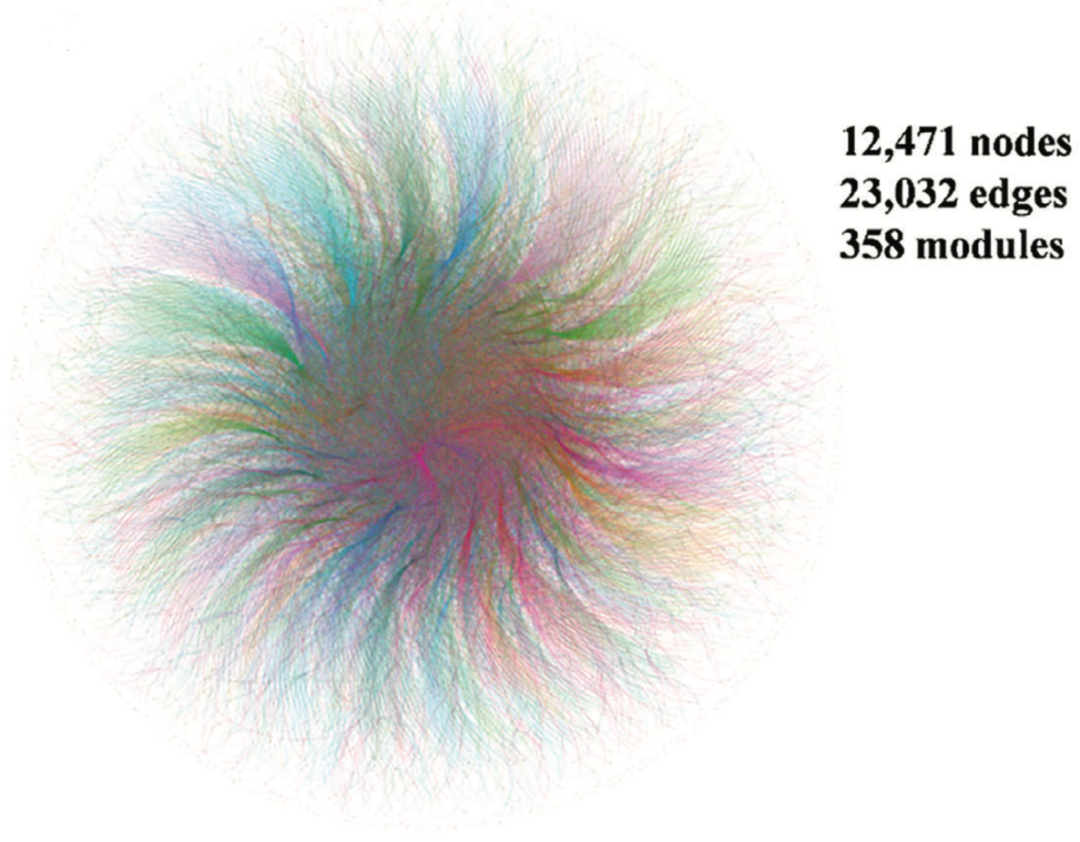
Fig4. The overall picture of RiPPID. The nodes represent proteins in RiPPID; the edges indicate PPI relationships; the modules indicate the nodes were classified in RiPPID with community detection. The various colors represent different modules
结论
1
该研究通过技术革新的 BIP-seq 不仅实现了高通量的水稻蛋白互作组的发现,而且研究团队通过对很多细节的改进实现了经济、高效的蛋白互作组的发现,极大扩展了现有的水稻蛋白互作组的覆盖范围。
2
另外,该技术还可用于其他物种内或物种间蛋白互作组的探索,具有极高的通用性及应用价值。
