
尽管人类和酵母在进化上存在数十亿年的分歧,但仍然共有数千个基因。虽然许多人类基因可以在功能上取代酵母对应的基因,但近一半的测试共享基因不能。例如,大多数酵母菌蛋白酶体亚基是除了包含 β 环核心的亚基,包括 β2c(HsPSMB7,一种组成性蛋白酶体亚基)。
我们开发了一个高通量的管道来人源化酵母蛋白酶体,通过生成一个大规模的 Hsβ2c 突变体文库,并筛选它们以补充酵母 β2(ScPup1)敲除。能够取代 ScPup1 的变异包括影响局部蛋白-蛋白相互作用(PPIs)的变异,其中影响 β2c C 端尾部与邻近 β3 亚基之间相互作用的变异最多 β2c 蛋白水解活性。将人类 β2c 的全长尾巴与 ScPup1 的全长尾巴交换,可以实现互补。此外,如果提供人 β3,野生型人 β2c 可以替代酵母 β2。出乎意料的是,酵母蛋白酶体具有催化活性阻断前体自动加工的 HsPSMB7-T44A 变体是可行的,这表明完整的前肽稳定了后期组装中间体。相比之下,类似的修饰在人 β2i (HsPSMB10) ,一个免疫蛋白酶体亚基和酵母 β2 的同源物,在酵母中不能实现互补,这表明在人免疫蛋白酶体核心组装中涉及不同的相互作用。总的来说,我们的数据揭示了特定 PPIs 在巨大进化距离中控制功能可替代性的作用。
一个新的高通量管道筛选酵母互补的人类基因变异
尽管作者之前用于识别酵母基因的人类基因替代型变异的筛选策略取得了成功,但该策略繁琐且仅鉴定出 2 个抑制子(Kachroo 等人,2015 年)。该技术需要人工分离菌落,然后进行四分体分离,而且鉴定抑制子需要人工筛选数百个酵母菌落。因此,作者开发了一种新的流程,以自动化高通量的方式筛选抑制子质粒(图 1a)。
作者特别专注于筛选非互补的人类 β2c(HsPSMB7)蛋白中的抑制子突变,这是一种组成型表达的蛋白酶体亚基。该方法能够在显著缩短的时间内实现大规模且无误的筛选。实验采用 96 孔板形式进行,并通过合成遗传阵列(SGA)或 MM 选择法分离单倍体特异性突变体,从而无需进行四分体分离(Pan 等人,2004 年;Kuzmin 等人,2016 年)。通过使用 5-FOA 选择的质粒依赖性测定,证实了互补作用与基于 URA3 的质粒上的变异人类基因相关。利用这一策略,作者成功获得了 HsPSMB7 基因中的多个可替换的抑制突变。在整篇论文中,作者始终使用“功能替换”、“可替换性”或“可替换能力”这些术语来指代人类基因或其变异体补偿其酵母同源基因功能的能力。该筛选流程是基于多项考虑因素而开发的。野生型人类 PSMB7(β2c)无法替代同源的酵母 β2 基因 ScPUP1,这一点从选择性剔除酵母基因时观察到的致死表型中可以得到证明(图 1b,补充图 1b)(卡赫罗等,2015 年)。相比之下,如果人类基因(或其变体)成功替代宿主基因的功能,该菌株将能够生长,从而作为功能替代的简单检测指标。错误率较高的 PCR 策略在带有 URA3 标记的酵母表达载体中生成了一个 HsPSMB7 突变基因库,每个基因平均有 1 至 4 个突变(图 1a)。
为了确定任何突变的 PSMB7 等位基因是否能够弥补删除同源酵母基因所导致的致死性,作者将突变基因库转化到酵母二倍体 HetKO PUP1/pup1Δ::kanMX 菌株中(Pan 等人,2004 年)。转化方案被扩展以获得数千个彼此分离的酵母菌落,每个菌落携带不同的 PSMB7 突变基因质粒(图 1b)。使用 QPix 460 以自动化方式选取了数千个菌落,并以 96 孔格式将其接种到预孢子化 GNA 培养基上,并通过 G418 选择法对带有 kanMX 标记的 pup1Δ 进行筛选。孢子形成后,在 MM 培养基(不含 Leu、Arg、His、Ura 和 CAN)中能够选择出携带不同人类 PSMB7 等位基因的可存活的 pup1Δ::kanMX 单倍体酵母孢子(在存在 G418 的情况下)(图 1c,底部面板)。作为孢子形成效率的内部对照,作者还测试了野生型 PUP1 单倍体孢子在 MM 培养基(不含 G418)上的生长情况(图 1c,顶部面板)。筛选结果显示,在 MM + G418 培养基上生长的 19 个菌落(图 1d 中的代表性图像)可能携带互补的人类 PSMB7 变体。然后对这些单倍体抑制突变体进行测试,以确定抑制是否是由于携带质粒中的人类突变基因所致。根据对 5-FOA 的缺乏生长(这会抑制 URA3 基因)来测试酵母细胞的质粒依赖性。这 19 个抑制突变体中有 7 个在 5-FOA 培养基上无法存活,这表明这些菌株中的人类基因变体对于其存活是必需的(图 1d)。
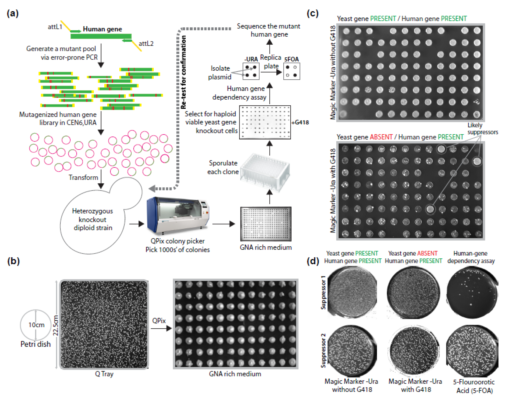
图 1:一种用于在酵母中筛选具有替代能力的人类基因抑制剂的高通量自动化流程。a) 工作流程展示了通过错配 PCR(每千碱基对 0 - 4 个突变)生成并筛选人类基因突变库的过程。变异池被克隆到表达载体(CEN6,URA3)中,并转化到酵母杂合二倍体敲除 PUP1/pup1Δ::kanMX 株中。b) 突变库的转化规模扩大(4 倍),混合物被涂布在 QTray 上。通过 QPix 460 菌落挑选机器人(最多 1000 个菌落)挑取单个菌落,并在富含 GNA 的预孢子形成培养基上点样,随后在 96 孔板格式中进行孢子形成。c) 每个孢子形成混合物被点样在含有 G418 的 MM 培养基上,表明“酵母基因缺失且人类基因存在”的条件,用于筛选具有替代能力的人类基因变体。或者,未添加 G418 的孢子形成混合物“酵母基因存在或缺失且人类基因存在”的条件,允许野生型单倍体酵母细胞生长,作为孢子形成效率的对照。为简便起见,我们将这些条件分别称为“含(+)G418”,以表示“酵母基因缺失”条件,以及“不含(-)G418”,以表示“酵母基因存在”条件。可能的抑制子表现为在含 G418 的 MM 培养基上生长的斑点,类似于不含 G418 的 MM 培养基条件下的生长情况。d)在含 G418 的 MM 培养基上生长的菌落(酵母基因缺失且人类基因存在条件)进一步通过使用 5-氟尿嘧啶(5-FOA)选择针对 URA3 质粒的质粒依赖性测定进行分析。代表性示例显示携带人类基因抑制子的酵母菌株在 5-FOA 培养基上不生长,表明其质粒依赖性(抑制子 1,上图),或在 5-FOA 培养基上生长,表明质粒独立性(抑制子 2,下图)。最后,对每个携带人类基因抑制子的质粒进行纯化并重新测试,以确认功能可替代性和质粒依赖性,随后进行 Sanger 测序以鉴定人类基因中的突变。
如果人 β3 也存在,野生型人 β2c 可以与酵母菌蛋白酶体结合
理想情况下,将酵母菌人源化以功能表征人类基因将利用酵母菌中的野生型人类等位基因。然而,抑制子筛选和 c 末端尾部交换数据表明,人类 β2c 需要突变与邻近的酵母亚基相互作用,特别是 Scβ3,才能正确地组装到酵母 CP 中。因此,恢复这些人类特异性亚基相互作用的策略可能使野生型 HsPSMB7 整合到酵母蛋白酶体中。为了验证这一假设,作者询问野生型人类 β2c 是否可以在功能上取代菌株中的酵母对应物。作者开发了一种基于 CRISPR-Cas9 的方法来靶向酵母的 ScPUP1 (β2) 和 ScPUP3 (β3) 基因。表达 Cas9-sgRNAScPUP1 或 Cas9-sgRNAScPUP3 的质粒转化酵母会导致其死亡。如果人类基因能够功能性地替代酵母的同源基因,那么将含有对应酵母位点 5' 和 3' 末端同源序列的人类基因修复模板进行共转化,预计会得到存活的细胞。
作者首先测试了野生型 HsPSMB7 是否能够替代 ScPUP1 在原位基因位点上发挥作用,但未能获得任何存活的菌落,正如预期的那样(图 2a)。然而,使用具有 T44A 或 S214G 突变的 HsPSMB7 变体,修复模板允许功能性地替换酵母的 PUP1 基因(图 2b)。此前,作者已经证明当酵母的 β3(PUP3)基因在质粒上表达时,它可以被其人类同源基因(HsPSMB3)功能性地替代(Kachroo 等人,2015 年)。使用基于 HDR 的 CRISPR-Cas9 策略(阿赫梅托夫等人,2018 年),作者成功地用人类的 β3(HsPSMB3)基因替换了解析型酵母的 β3 基因(图 2c)。因此,酵母的 β2 亚基能够将人类的 β3 亚基招募至酵母蛋白酶体核心部位,但人类的 β2c 亚基却无法与酵母的 β3 亚基共同发挥这一作用。首先使用了人化的 β3 酵母菌株,基于 CRISPR-Cas9 的基因编辑技术现在能够将酵母中的 β2 替换为野生型的人类 β2c(HsPSMB7)基因。双人化的 Hsβ2c-Hsβ3 酵母菌株是可存活的,这一点通过位点特异性 PCR 和桑格测序得到了验证(图 2d)。
因此,通过提供其相邻的人类亚基,即人类 β3,通常不互补的人类 β2c 现在能够在酵母蛋白酶体中发挥作用。然而,在酵母中提供人类 β3 并不能使酵母 β2 通过 Hsβ2i 获得互补。定量生长测定显示了工程菌株的适度适应性缺陷。人化的 Hsβ2c-S214G 菌株在 23°C 时表现出低温敏感的生长缺陷,而人化的 Hsβ2c-T44A 菌株在 23°C 和 37°C 时的生长速度比野生型菌株慢。此外,虽然人化的 Hsβ3 菌株与野生型酵母生长情况相当,但人化的 Hsβ2c-Hsβ3 菌株在 23°C 时表现出低温敏感的表型,并且在液体培养中在 30°C 时生长速度较慢。
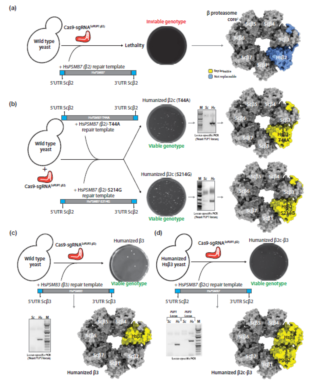
图 2:同时提供野生型的人类 β2c 和相邻的人类 β3 能够实现其酵母同源物的功能替代。a) 将 pCas9-sgRNAScPUP1 和人类野生型 PSMB7 基因修复模板作为 PCR 片段进行共转化,无法获得存活的人源化菌株。b) 然而,将 pCas9-sgRNAScPUP1 和人类 PSMB7-T44A 或 PSMB7-S214G 基因变体作为修复模板进行共转化,可获得基因组整合了人类基因变体的存活酵母。c) 将 pCas9-sgRNAScPUP3 和人类野生型PSMB3(β3)基因修复模板作为 PCR 片段进行转化,可获得存活的人源化 β3 菌株。d) 以人源化 β3 菌株为背景,将 pCas9-sgRNAScPUP1 和人类野生型 PSMB7 基因作为修复模板进行共转化,可获得基因组整合了野生型人类 β2-β3 的酵母。使用 ChimeraX 软件展示了单个由 7 个亚基组成的酵母 β - 蛋白酶体核心环(PDB-1RYP)。可替代的亚基以黄色显示,不可替代的亚基以蓝色显示。代表性培养皿显示在 30°C 培养 3 - 5 天后在选择培养基上生长的菌落。

